
1
Course: Medical Microbiology
Lecturer: Dr. Weam Saad
Subject: Medical Bacteriology
Medical Bacteriology
Anatomy of bacteria
All bacteria, either pathogenic or saprophytic, are unicellular organisms
that reproduce by binary fission. Most bacteria are capable of independent
metabolic existence and growth, but species of Chlamydia and Rickettsia are
obligate intracellular organisms. Bacterial cells are extremely small and
measured in microns (10-6µm). They range in size from large cells such as
Bacillus anthracis (1.0 to 1.3 µm X 3 to 10 µm) to very small cells such as
Pasteurella tularensis (0.2 X 0.2 to 0.7 µm), Mycoplasmas (atypical pneumonia
group) are even smaller, measuring 0.1 to 0.2 µm in diameter.
Bacteria have characteristic shapes. The common microscopic
morphologies are cocci (round cells, such as Staphylococcus aureus or
Streptococcus sp.; rods, such as Bacillus and Clostridium species; long,
filamentous branched cells, such as Actinomyces species; and comma-shaped
and spiral cells, such as Vibrio cholerae and Treponema pallidum.
The arrangement of cells is also typical of various species or groups of
bacteria. Some rods or cocci characteristically grow in chains; some, such as
Staphylococcus aureus, form grapelike clusters of spherical cells; some round
cocci form cubic packets. Bacterial cells of other species grow separately. The
microscopic appearance is important in classification and diagnosis.

2
Surface Appendages
Two types of surface appendage can be recognized: Flagella, which are
organs of movement, and Pili (in Latin =hairs), which are also known as
fimbriae ( in Latin = fringes).
Flagella occur on both Gram-positive and Gram-negative bacteria, and their
presence can be useful in identification. For example, they are found on many
species of bacilli but rarely on cocci. In contrast, pili occur almost on all Gram-
negative bacteria and are found on only a few Gram-positive organisms (e.g.,
Corynebacterium renale). Some bacteria have both flagella and pili. The
electron micrograph in Fig. below shows the characteristic wavy appearance of
flagella and two types of pili on the surface of Escherichia coli.

3
1. Flagella
Bacterial flagella are long (3 to 12 µm), filamentous surface appendages
about 12 to 30 nm in diameter. A flagellum consists of three parts:
(1) The long filament, which lies external to the cell surface.
(2) The hook structure at the end of the filament.
(3) The basal body, to which the hook is anchored and which imparts
motion to the flagellum.
The ability of bacteria to swim by flagella provides them with the
mechanical means to undergo chemotaxis (movement in response to attractant
and repellent substances in the environment).
Chemically, flagella are constructed of a class of proteins called flagellins.
Flagellins are immunogenic, these antigens are called the H antigens, which are
characteristic of a species or strain of bacteria. The species specificity of the
flagellins reflects differences in the primary structures of the proteins. Antigenic
changes of the flagella known as the phase variation of H1 and H2 occurs in
Salmonella typhimurium. The number of flagella on bacterial surface is a
characteristic for classification. Flagella formation can be inhibited by
chloramphenicol it blocks regeneration of flagella and the protein (flagellin)
synthesis.
Figure below illustrates typical arrangements of flagella on or around the
bacterial surface. For example, V. cholerae has a single flagellum at one pole of
the cell (i.e., it is monotrichous), whereas Proteus vulgaris and E. coli have
many flagella distributed over the entire cell surface (i.e., they are peritrichous).
The flagella of a peritrichous bacterium must aggregate as a posterior bundle to
propel the cell in a forward direction.

4
2. Pili
The terms pili and fimbriae are usually used to describe the thin, hairlike
appendages on the surface of many Gram-negative bacteria and proteins of pili
are referred to the pilins. Pili (plural of pilus) are more rigid than flagella. In
some bacteria, such as Shigella species and E coli, pili are distributed over the
cell surface as 200 per cell.
As in E coli, pili can come in two types: short, most common pili, and sex
pili
(one to six of very long pili). The sex pili attach male to female bacteria
during conjugation.
Pili in many enteric bacteria offer the adhesive properties on the bacterial
cells, enabling them to adhere to various epithelial surfaces and to the RBCs
(causing hemagglutination). These adhesive properties play an important role in
bacterial pathogenesis; colonization of epithelial surfaces and are therefore
called colonization factors.

5
Surface Layers
1. Capsules and Loose Slime
Some bacteria form capsules, they are thick layer of viscous gel. Capsules
may be up to 10 µm thick. Some organisms lack a well-defined capsule but
have loose slime layers external to the cell wall or cell envelope. The hemolytic
Streptococcus mutans, the primary organism found in dental plaque is able to
synthesis a large extracellular mucoid glucans from sucrose.
Not all bacterial species produce capsules; the capsules of encapsulated
pathogens are often important determinants of virulence. In both groups, Gram-
positive and Gram-negative bacteria; most capsules are composed of high
molecular-weight polysaccharides outside the cell wall or envelope except the
capsule of Bacillus anthracis (the pathogen of anthrax), it is unusual in that it is
composed of a g-glutamyl polypeptide. Mutation can cause loss of enzymes
involved in the biosynthesis of the capsular polysaccharides can result change
from the smooth-to-rough as seen in the pneumococci.
The exact function of capsule is resistance to phagocytosis and protection
of the bacterial cell against the host defenses during invasion. Some bacterial
capsules work as main virulence factor.
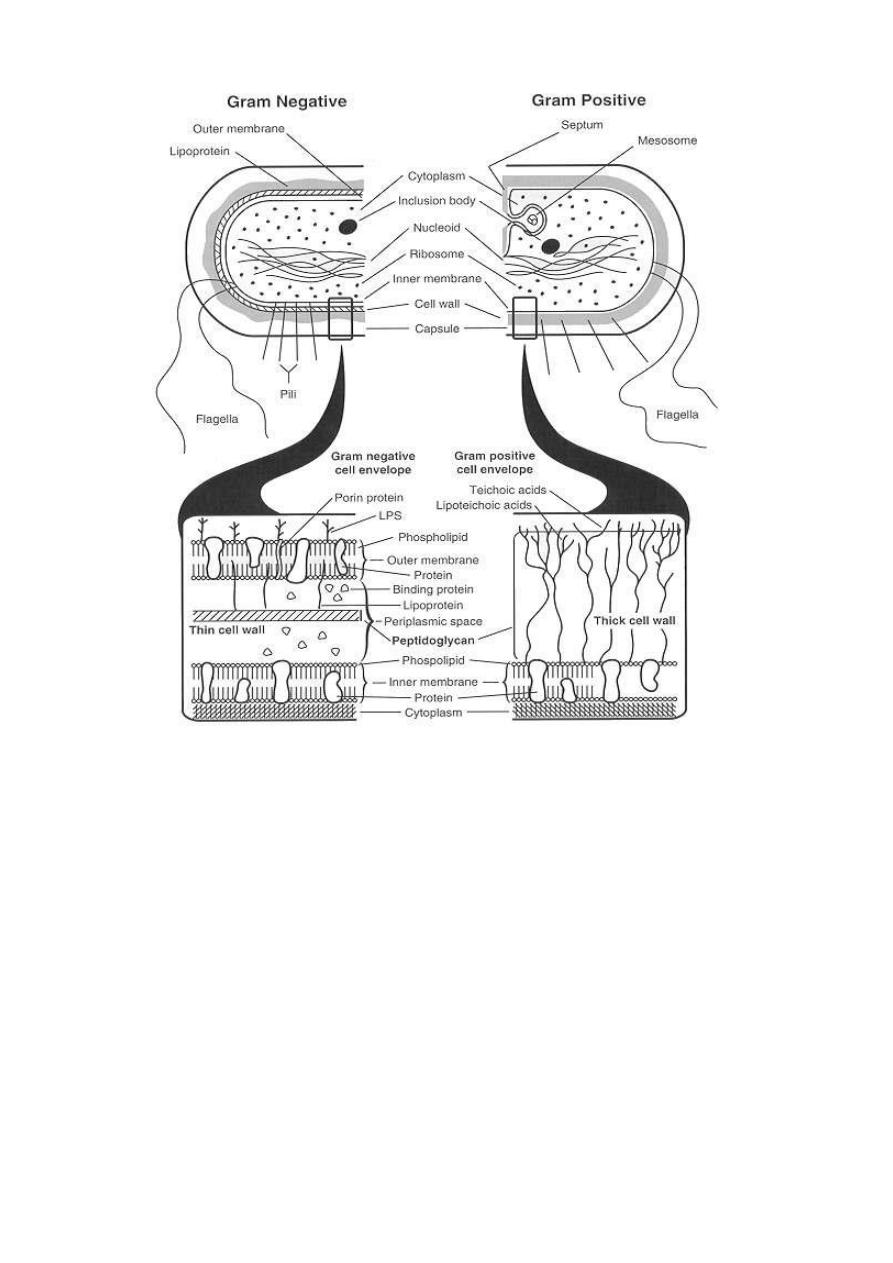
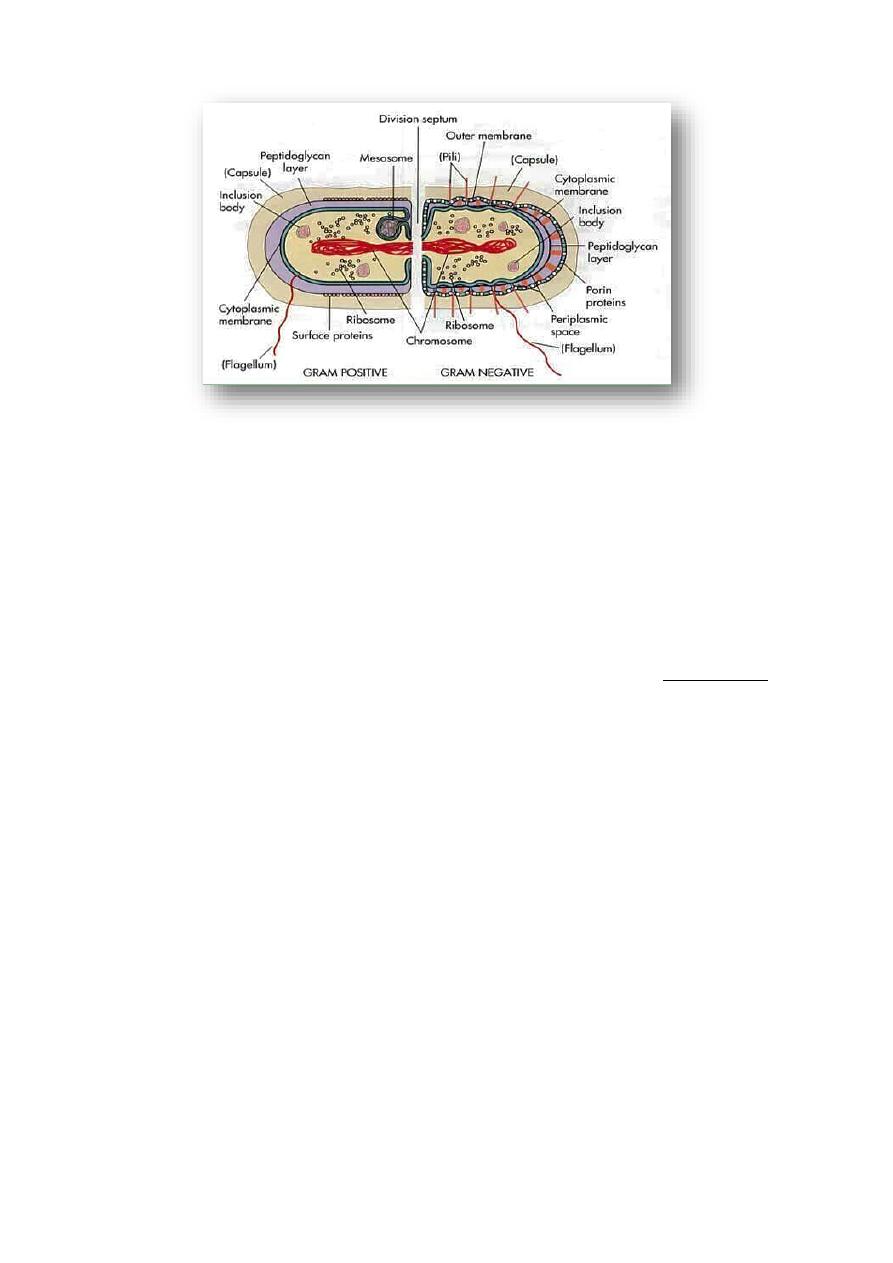
2. Cell Wall
The Gram stain differentiates bacteria into Gram-positive and Gram-
negative groups. Gram-positive and Gram-negative organisms differ in the
structures of their cell walls, see figure below.
Most Gram-positive bacteria have a thick (about 20 to 80 nm), cell wall
composed of peptidoglycan (also known as mucopeptide or murein). In thick
cell walls, some other cells have other wall polymers (such as the teichoic acids,
polysaccharides, and peptidoglycolipids) are attached to the peptidoglycan. The
peptidoglycan layer in Gram-negative bacteria is thin (about 5 to 10 nm thick).
The basic differences in surface structures of Gram-positive and Gram-
negative bacteria explain the results of Gram staining.
6
Peptidoglycan
Unique composition in all prokaryotic cells (except for mycoplasmas) is
the peptidoglycan, this layer help in mechanical protection and there are
specific enzymes involved in its biosynthesis. These enzymes are target sites for
inhibition of peptidoglycan synthesis by specific antibiotics. The primary
chemical structures of peptidoglycans consist of a polymer backbone of
disaccharides of N-acetylmuramyl-N-acetylglucosamine and are linked through
the carboxyl group by amide linkage of muramic acid residues.
There are two groups of bacteria that lack the peptidoglycan, the
Mycoplasma species (causes atypical pneumonia and some genitourinary tract
7
infections) and the L-forms. The mycoplasmas and L-forms are all Gram-
negative and insensitive to penicillin.
Teichoic Acids
The teichoic acids are found only in some Gram-positive bacteria (such as
Staphylococci, Streptococci, Lactobacilli, and Bacillus spp); they are not found
in gram- negative bacteria. Teichoic acids are polyol phosphate polymers, with
either ribitol or glycerol linked by phosphodiester bonds. It can act as a specific
antigenic determinant.
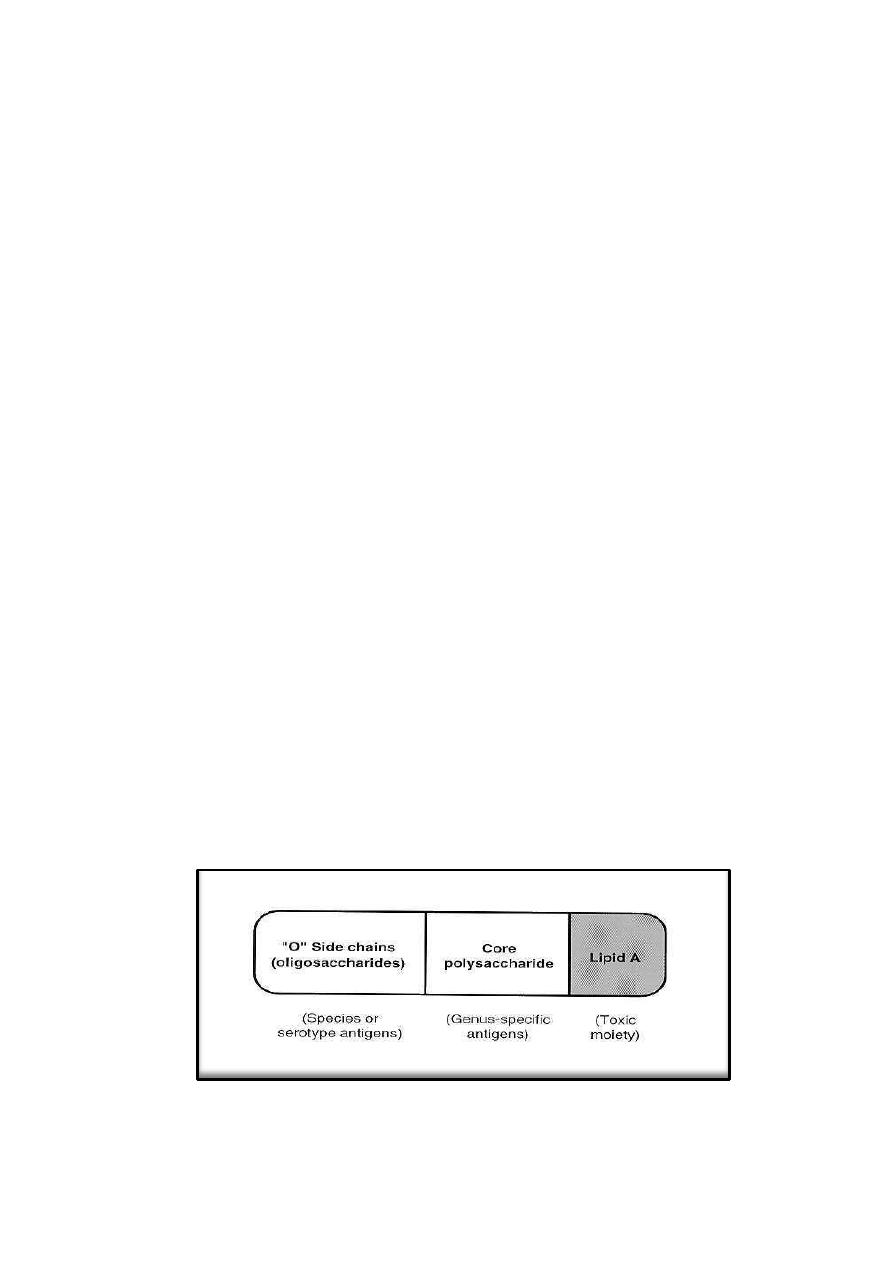
Lipopolysaccharides
A characteristic of Gram-negative bacteria is the lipopolysaccharide (LPS),
only one Gram-positive organism, Listeria monocytogenes, has been found to
contain LPS. The LPS are also called endotoxins, they are cell-bound, heat-
stable toxins and differ from heat-labile, protein exotoxins secreted into culture
media. Endotoxins possess an array of powerful biologic activities and play an
important role in the pathogenesis of many Gram-negative bacterial infections.
In addition LPS is pyrogenic and causes endotoxic shock, can activate
macrophages and complement system, it is mitogenic for B lymphocytes,
induces interferon production, causes tissue necrosis and tumor regression, and
has adjuvant properties. The endotoxic properties of LPS is due to the lipid A
components. Usually, the LPS molecules have three regions: The lipid A
attached to the core composed of polysaccharide chains which are linked to the
O-antigens responsible for serologic specificity of the Gram-negative bacteria.
8
Intracellular Components
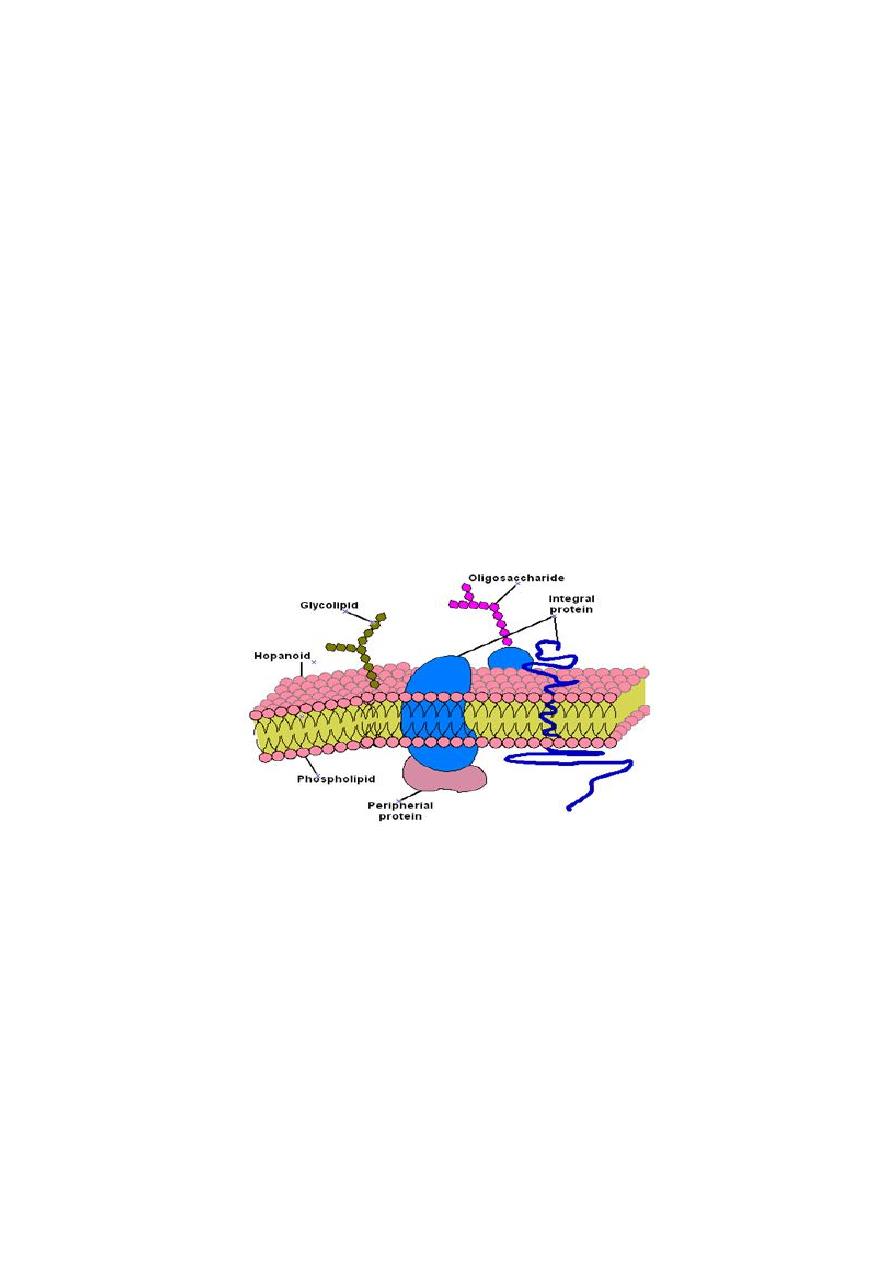
1. Plasma (Cytoplasmic) Membranes
Bacterial plasma membranes is similar to eukaryotic plasma membranes in
function, are referred to cytoplasmic or protoplast membranes, they are
composed primarily of proteins and lipids (phospholipids). Protein-to-lipid
ratios of bacterial plasma membranes are approximately 3:1, close to those for
mitochondrial membrane.
Plasma membranes are the site of active transport, respiratory chain
components, energy-transducing systems, the ATPase of the proton pump, and
membrane stages in the biosynthesis of phospholipids, peptidoglycan, LPS, and
capsular polysaccharides. The bacterial cytoplasmic membrane is a
multifunction structure similar to mitochondrial transport and biosynthetic
functions of eukaryotic cells. The plasma membrane is also the anchoring site
for the bacterial DNA.
2. Mesosomes
The mesosomes are tubular-vesicular membrane structures found in Gram-
positive bacteria which are formed by an invagination of the plasma membrane.
These structures equivalent to bacterial mitochondria; and may be related to
events in the cell division cycle.
9
3. Other Intracellular Components
Ribosomes of the 70S type; ribonucleoprotein particles are not arranged on a
membranous rough endoplasmic reticulum as they are in eukaryotic cells, they
are found in the cytoplasm.
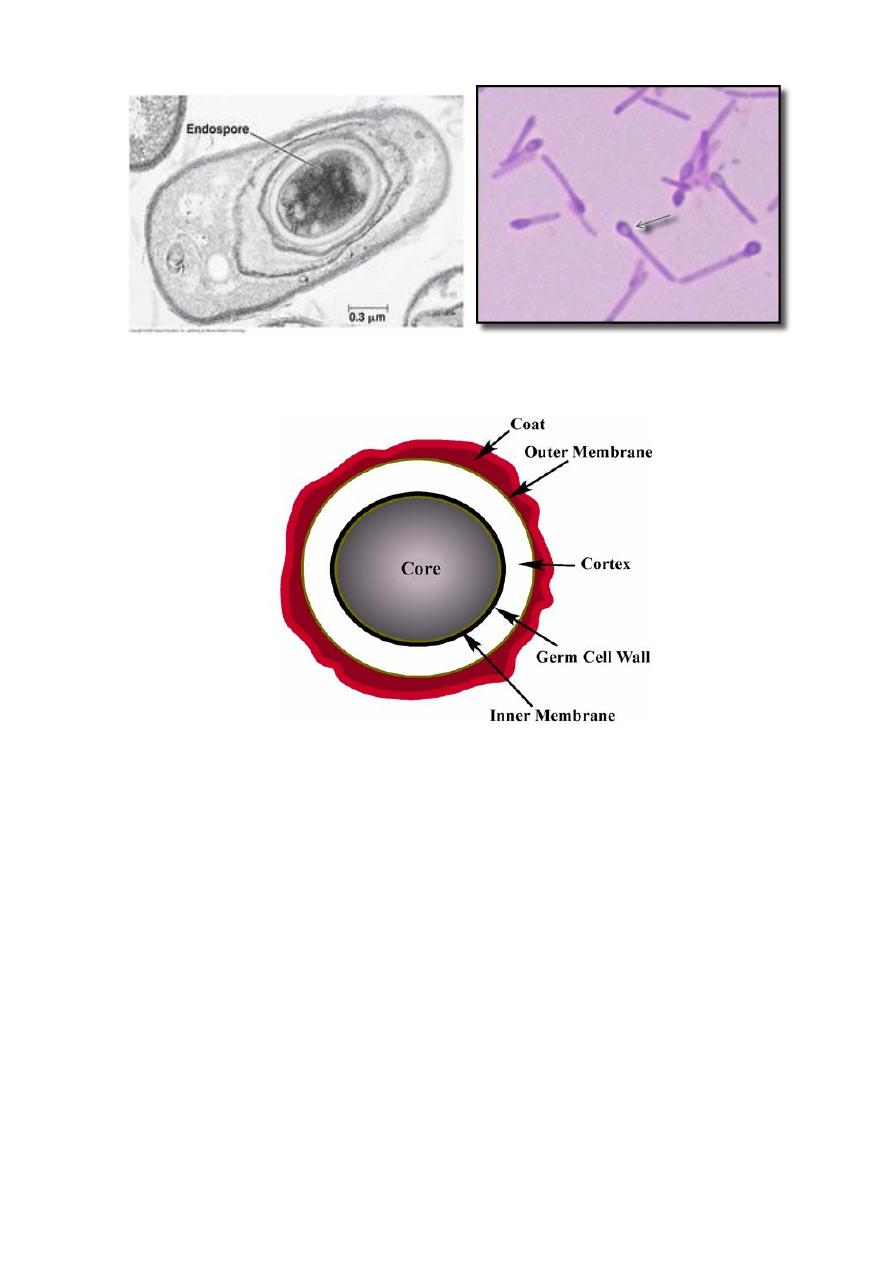
Endospores are highly heat-resistant, dehydrated cells formed
intracellularly in some bacteria like Bacillus and Clostridium. Sporulation, is
the process of forming endospores because of many biochemical and
morphologic changes begins in the stationary phase of the vegetative cell cycle
due to decrease nutrients (sources of carbon and nitrogen). Also formation of
unusual peptidoglycan which contains calcium dipicolinate, help in resistance
to heat, radiation, pressure, and organic solvents.
The spore protoplast, or core, contains a complete chromosome,
ribosomes, and energy generating components. During germination, the spore
wall becomes the vegetative cell wall and cortex will be released.
10
Genetic Information In Bacteria (Genome)
Genetic material, genome or the genotype replicate in the parental organism
to give the daughter organism a copy of the genetic material. Expression of
genetic material determines the observable characteristics (phenotype) of the
organism. Some bacteria have the DNA chromosome as the only genetic
material (genome) in bacterial cells, like Escherichia coli. Other bacteria have
additional genetic materials, such as plasmids and bacteriophages.

11
1. Chromosomal DNA
Bacterial genomes differ in size from about 0.4 x 109 to 8.6 x 109 daltons
(Da), some of the smallest found in the obligate parasites (Mycoplasma) and the
largest in Myxococcus. The amount of DNA in the genome determines the
maximum amount of information that it can encode. Most bacteria have a
haploid genome, a single chromosome consisting of a circular, double stranded
DNA molecule. However linear chromosomes have been found in Gram-
positive Borrelia and Streptomyces spp.,
The typical genome to be studied is the E coli genome, it is sufficient to
code for thousand polypeptides of average size (40 kDa or 360 amino acids),
the DNA is supercoiled and tightly packaged in the bacterial nucleoid. The
time required for replication of the entire chromosome is about 40 minutes,
which is approximately twice the shortest division time for this bacterium.
The replication of chromosomal DNA in bacteria is complex and involves
many different proteins, in rapidly growing bacteria a new round of
chromosomal replication begins before an earlier round is completed. Thus, the
chromosome is replicating at more than one point. Bacterial chromatin does not
contain basic histone proteins, but low-molecular-weight polyamines and
magnesium ions may give a function similar to that of eukaryotic histones.
2. Plasmids
Plasmids are extrachromosomal genetic elements in bacteria. They are
smaller than the bacterial chromosome. Plasmids usually encode properties that
are not essential for bacterial viability, and replicate independently of the
chromosome.
Most plasmids are supercoiled, circular, double-stranded DNA molecules,
The Large plasmids (Conjugative plasmids) promote transfer of the bacterial
chromosome from the donor bacterium to other recipient bacteria are also called
fertility plasmids. The small plasmids are usually non-conjugative.
Many plasmids control medically important properties of pathogenic
bacteria, including resistance to antibiotics, production of toxins, and synthesis
of cell surface structures required for adherence or colonization. Plasmids that
12
determine resistance to antibiotics are called R plasmids (or R factors) like the
plasmid in Staphylococcus aureus. Some toxins encoded by plasmids include
heat-labile and heat-stable enterotoxins of E. coli, exfoliative toxin of
Staphylococcus aureus, and tetanus toxin of Clostridium tetani. Some plasmids
have no recognizable effects on the bacterial cells that have them.
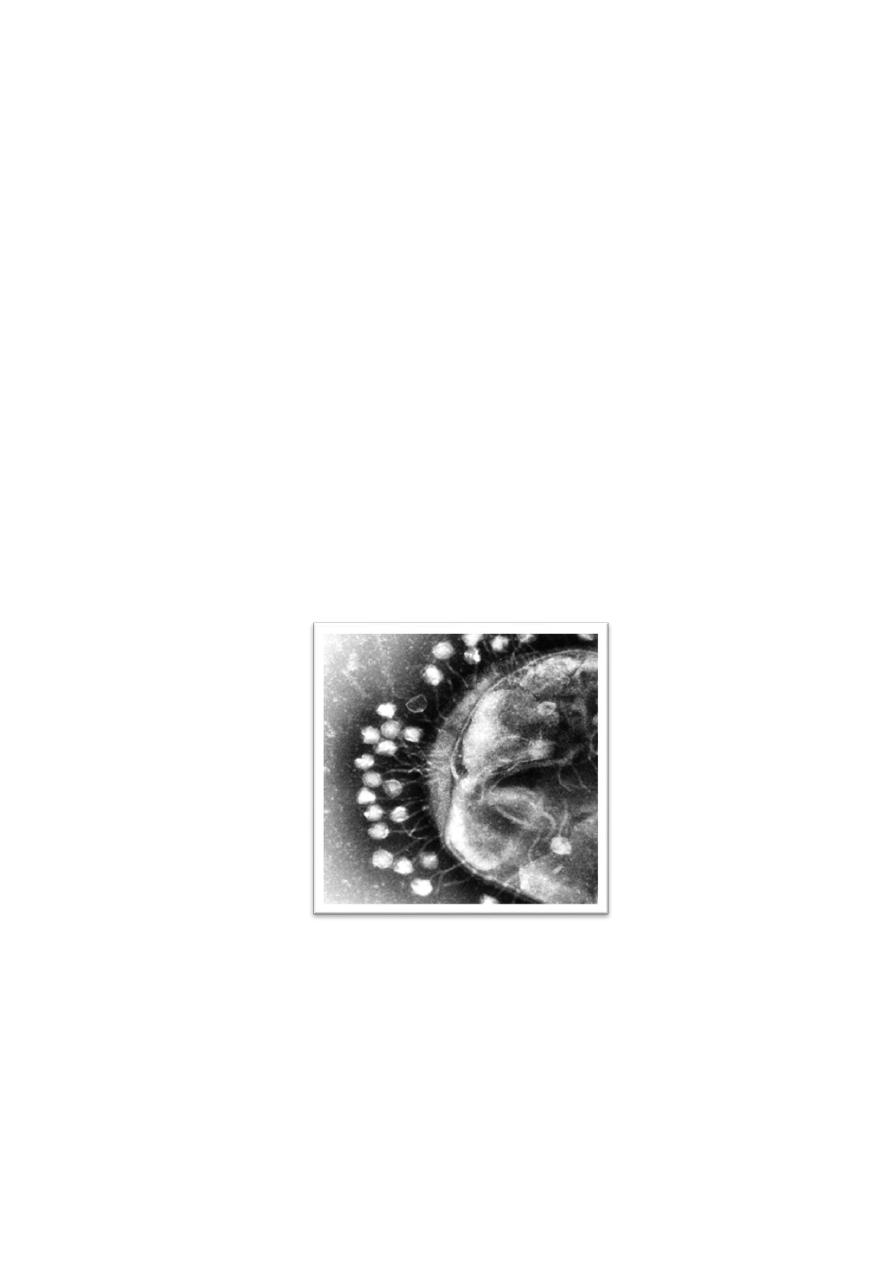
3. Bacteriophages
Bacteriophages (bacterial viruses, phages, viruses that infect bacteria) are
infectious agents for bacteria that live as obligate intracellular parasites inside
bacteria, consist proteins plus nucleic acid (DNA or RNA, but not both). The
proteins of the phage particle form a protective shell (capsid) surrounding the
tightly packaged nucleic acid genome.
Phage genomes are different in size, consist of double-stranded DNA,
single-stranded DNA, or RNA. Phage genomes, like plasmids, encode functions
required for the replication of virus in bacteria, they also encode capsid proteins
and nonstructural proteins required for phage life cycle.
Bacteriophages attached on bacteria surface under electron
microscope
For example of phages responsible of virulence: the production of
diphtheria toxin by Corynebacterium diphtheriae, erythrogenic toxin by
Streptococcus pyogenes (group A beta-hemolytic streptococci), botulinum toxin
by Clostridium botulinum, and Shiga-like toxins by E. coli.

13
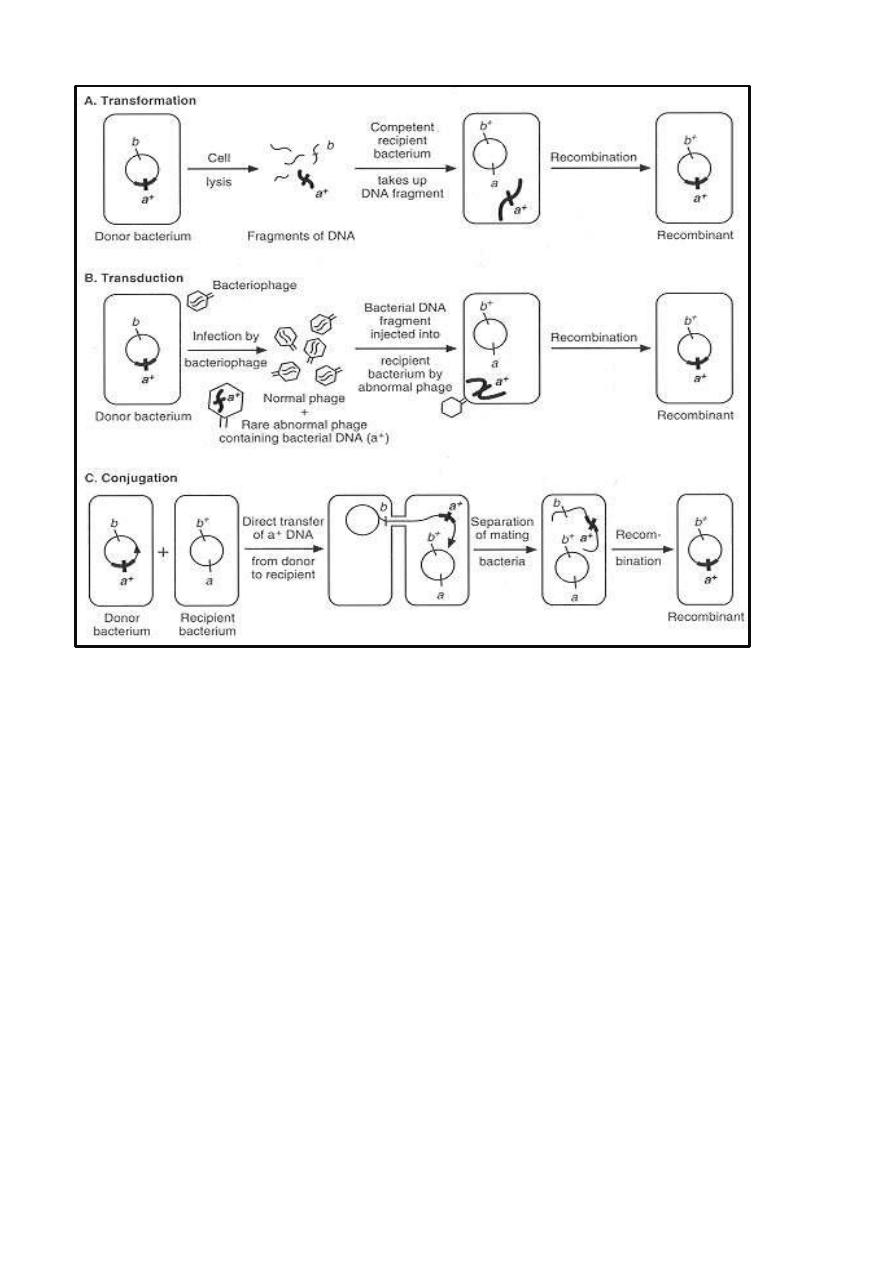
Exchange of Genetic Information
A phenomena of medical importance that involve exchanges of genetic
information or genomic rearrangements include the rapid antibiotic resistance
plasmids, flagellar phase variation in some bacteria like Salmonella, and
antigenic variation of surface antigens like in Neisseria and Borrelia. These
processes involve Transformation, transduction, and conjugation
1. Transformation
In transformation, pieces of DNA (at least 500 nucleotides in length)
released from donor bacteria are taken up directly from the extracellular
environment by other recipient bacteria. Recombination occurs between single
molecules of transforming DNA and the chromosomes of recipient bacteria.
Transformation was discovered in Streptococcus pneumoniae and occurs in
other bacterial genera including Haemophilus, Neisseria, Bacillus, and
Staphylococcus.
2. Transduction
In transduction, bacteriophages function as vectors to carry a small segment
of the bacterial genome (DNA genes) from donor bacteria into other recipient
bacteria by infection. When phage infects a recipient cell, expression of the
transferred donor genes occurs and complete transduction is characterized by
production of new proteins (donor phenotype). In abortive transduction the
donor DNA fragment does not replicate.
3. Conjugation
In conjugation, direct contact between the donor and recipient bacteria leads
to establishment of a cytoplasmic bridge between them and transfer of part or
all of the donor genome to the recipient. Donor bacteria must have conjugative
plasmids called fertility+ plasmids, sex plasmids or F factor, e.g. F plasmid of
E. coli.
14
Recombination DNA and Gene Cloning (Genetic Engineering)
Recombination involves breakage and joining of DNA molecules to form
hybrid, recombinant molecules. Several kinds of recombination have been
identified by the help of specific enzymes that act on DNA (e.g., exonucleases,
endonucleases, polymerases, ligases) participate in recombination .
Many methods are available to make hybrid DNA molecules in vitro
(recombinant DNA). Cloned genes can be expressed in appropriate host cells,
and the phenotypes that they show can be determined.
Applications of DNA cloning are expanding rapidly in all fields of
biology and medicine :

15
In medical genetics such applications range from the prenatal diagnosis of
inherited human diseases to the characterization of oncogenes and their
roles in carcinogenesis .
Pharmaceutical applications include production from cloned human genes
of biologic products with therapeutic purposes, such as hormones,
interleukins, and enzymes .
Applications in public health and laboratory medicine include
development of vaccines to prevent specific infections by polymerase
chain reaction (PCR). The PCR process uses oligonucleotide primers and
DNA polymerase to amplify specific target DNA sequences during
synthesis in vitro, help to detect target DNA sequences in clinical
specimens with great sensitivity.
